|
|
| (No se muestra una edición intermedia de un usuario) |
| Línea 4: |
Línea 4: |
| | | | |
| | A continuación se expondrá brevemente la irrupción histórica del paradigma epigenético. Después, procederemos a una descripción básica de la estructura y de la función del material genético que posibilite entender los principales mecanismos de regulación de la expresión génica. Esto permitirá obtener una visión general del significado de la dimensión epigenética del desarrollo. | | A continuación se expondrá brevemente la irrupción histórica del paradigma epigenético. Después, procederemos a una descripción básica de la estructura y de la función del material genético que posibilite entender los principales mecanismos de regulación de la expresión génica. Esto permitirá obtener una visión general del significado de la dimensión epigenética del desarrollo. |
| − |
| |
| − |
| |
| − | ==La irrupción del paradigma epigenético==
| |
| − |
| |
| − | Históricamente, la palabra "epigenética" fue utilizada para describir eventos que no podían ser explicados por los principios genéticos. Conrad Waddington (1905-1975), considerado el primero en acuñar el término, definió la epigenética como "la rama de la biología que estudia las interacciones causales entre los genes y sus productos que dan lugar al fenotipo” (Waddington 1942). En 1957, Waddington propuso el concepto de “paisaje epigenético” en una conocida metáfora visual dinámica para representar el proceso de diferenciación celular. La célula, representada por una pelota, se situaba en un paisaje montañoso donde podía tomar trayectorias específicas distintas dando lugar a diferentes resultados o destinos celulares (cfr. Goldberg et al. 2007)
| |
| − |
| |
| − | Con respecto a la importancia de la influencia epigenética en el desarrollo biológico encontrábamos desde mediados del siglo pasado cierta controversia. La discusión se producía, a grandes rasgos, entre aquellos que entendían al genoma como máximo responsable del desarrollo del organismo y aquellos que defendían un modelo explicativo epigenético como lo esencial del desarrollo biológico.
| |
| − |
| |
| − | Esta discusión está recogida de forma sintética y significativa en las publicaciones especializadas de referencia en embriología de mediados del siglo XX. Tras el descubrimiento de la bioquímica el ADN y su relación con los genes por Oswal Avery (Avery, MacLeod y Maclyn 1944), y justo antes del descubrimiento de su estructura en doble hélice por Watson y Crick (1953), en el apartado titulado “Epigénesis y preformación” del manual de referencia ''Human embryology'' firmado en 1945 por Hamilton, Boyd y Mossman se afirmaba que “la investigación en embriología durante los últimos cien años había demostrado de forma concluyente que el proceso de desarrollo era de naturaleza epigenética, pero que, por aquel entonces, la doctrina de la preformación había sido reintroducida a través de la explicación de los hechos establecidos por la genética moderna.”'' ''(Hamilton, Boyd y Mossman 1945, 4)''.'' En el mismo capítulo de la tercera edición revisada en 1964 (y también recogido íntegramente en la cuarta edición del año 1972), y tras dos décadas de estudios moleculares, los autores se inclinan por una postura “preformacionista” y reconocen, tímida pero claramente, un vacío que será cubierto, décadas después, por la epigenética tal y como ahora la conocemos:
| |
| − |
| |
| − | ''“La investigación en embriología durante los últimos cien años ha demostrado que el proceso de desarrollo es de naturaleza epigenética (…) sin embargo, la genética moderna ha mostrado que los genes localizados en los cromosomas del núcleo del cigoto (el huevo fertilizado), contienen la información necesaria que permite que ocurra el desarrollo normal. La constitución hereditaria de un individuo está determinada por el complemento de genes presente en los cromosomas y en este sentido, la ontogenia consiste esencialmente en la revelación gradual del plan almacenado en el genoma. Por tanto, la opinión actual sobre la herencia y constitución del organismo es rigurosamente pre-formacionista. Cómo y cuándo entran en juego los diferentes genes en el proceso de desarrollo no está todavía establecido”.'' (Hamilton, Boyd y Mossman 1964, 6; 1972, 7)
| |
| − |
| |
| − | Durante las más de cinco décadas que han transcurrido desde esta visión se han ido desvelando gradualmente las incógnitas del “cómo y cuándo” apuntado por Hamilton, Boyd y Bossman. En los últimos años se ha revelado que, en gran medida, la información almacenada en el genoma está regulada por factores de origen epigenético (Latschman 2010, 273).'' ''Atendiendo al desarrollo del embrión preimplantatorio, la regulación epigenética se presenta como la máxima responsable del desarrollo y especialización celular. En esta línea se sitúa el paradigma epigenético, que se ha ido consolidando avivado por una crítica hacia actitudes “fijistas” que entendían de forma simplista que en eucariotas toda la información está contenida en los genes desde el cigoto y no podía ser afectada por factores, por ejemplo, ambientales [https://kplumbinginc.com/ royaltoto].
| |
| − |
| |
| − | En la primera década del siglo XX la comunidad científica no cuestionó la necesidad e importancia de conocer e investigar la epigenia para explicar completa y correctamente la dinámica celular, y ya se afirma que el desarrollo del embrión en los mamíferos es, por definición, epigenético. Pero no solo eso: actualmente la constatación de la estabilidad parcial epigenética en las células somáticas y de línea germinal (Reik 2007) permite conocer los mecanismos moleculares de la herencia y puede proporcionar un nuevo marco para la búsqueda de factores etiológicos en rasgos complejos y enfermedades (Petronis 2010).
| |
| − |
| |
| − | Aunque la epigenética es un paradigma consolidado firmemente y asumido por la comunidad científica en los últimos años, es necesario matizar qué significado tiene el término “esencial” cuando este, desde una perspectiva científica y sin ninguna precisión semántica, se aplica a la epigénesis. Aparecen discusiones a este respecto que ofrecen una idea de la necesidad de una reflexión acerca de la comprensión del papel de la epigenética dentro de reflexiones interdisciplinares, pero también dentro de la propia comunidad científica.
| |
| − |
| |
| − | Un claro ejemplo de estas discusiones en el interior de la comunidad científica gira en torno al Consorcio Internacional del Epigenoma Humano (IHEC). La universalidad de los fundamentos de la regulación génica en todos los organismos y tipos celulares ha animado a parte de la comunidad científica internacional a sacar adelante el IHEC, cuyo objetivo es crear un mapa genómico que recoja las modificaciones epigenéticas y, con ellas, la variabilidad de la expresión génica con el objetivo principal de ayudar a los biólogos a comprender cómo la epigenética influye en la salud y la enfermedad. Las bases que fundamentan este objetivo crearon un controvertido debate dentro de la comunidad científica internacional. En el 2010, un artículo firmado por la Editorial de la revista ''Nature'' titulado “''Time for epigenome''” afirmaba, en clave de apoyo al IHEC, que los proyectos que se centraban en el genoma a gran escala ya indicaban que las secuencias de genoma, intra e interespecífico, eran demasiado similares como para ser capaces de explicar la diversidad de la vida, que se tornaba evidente que la epigenética podría explicar mejor cómo esos códigos genéticos similares se expresan de manera única en diferentes células, en diferentes condiciones ambientales y en diferentes momentos (Editorial 2010). Ante estas afirmaciones, varios científicos lanzaron duras críticas afirmando que aquella opinión parece no tener en cuenta los principios de la regulación génica y de la biología evolutiva y del desarrollo que se han establecido durante los últimos 50 años. Según explican, las marcas epigenéticas son dependientes directamente de la secuencia genómica y, por lo tanto, han de entenderse como los efectos de las interacciones reguladoras de secuencias específicas y no como las causas de la expresión génica específica de tipos celulares (Ptashne, Hobert y Davidson 2010). Eminentes científicos ya habían tratado con detalle esta cuestión redactando una carta, a la que se asociaron otros muchos expertos de todo el mundo, donde se exponían las serias reservas sobre la base científica del proyecto epigenoma (Madhani et al. 2008).
| |
| − |
| |
| − | La irrupción de la epigenética también ha supuesto un desafío para la biología evolutiva y la filosofía de la biología. Uno de los debates teórico-filosóficos más importantes se pregunta si la selección natural, por el carácter hereditario de la epigenética, opera en los cambios inducidos y adquiridos por un organismo, y analiza las implicaciones filosóficas y sociales de una extensión de la nueva síntesis o teoría sintética de la evolución (Jablonka y Lamb 2014; Dickens y Rahman 2012). El impacto de la epigenética se observa también en el surgimiento de la biología evolutiva del desarrollo (o informalmente evo-devo, del inglés ''evolutionary developmental biology''). La evo-devo es una disciplina de la biología evolutiva que compara el proceso de desarrollo de diferentes organismos con el fin de determinar sus relaciones filogenéticas. Inicialmente, desde este campo se buscaba identificar los mecanismos del desarrollo que dan origen a cambios evolutivos en los fenotipos de los individuos (Hall 2003), pero paulatinamente la evo-devo ha sufrido una enorme transformación debido, justamente, a su amplio espectro de intereses que involucran actualmente a más de una decena de disciplinas (cfr. Moczec et al. 2015, Nuño de la Rosa 2016, véase la voz Evo-devo en esta enciclopedia). La denominada eco-evo-devo (del inglés ''ecological evolutionary developmental biology'') es otro buen ejemplo a traer a colación. Se trata de una rama de la biología centrada en el estudio de los efectos de las señales ambientales en la generación de los fenotipos durante el desarrollo de un organismo, y de la influencia de esta afectación en el proceso evolutivo de las especies (Cfr. Gilbert, Bosch y Ledón-Rettig 2015). Cabe también destacar la discusión en torno a la noción de causalidad que los descubrimientos en torno a la epigenética han generado en el marco de la filosofía de la biología (Robert 2008).
| |
| − |
| |
| − | La bioética es otro contexto interdisciplinar que no ha sido ajeno a la aparición de la epigenética, y es en las discusiones en torno al estatuto ontológico del embrión humano donde la epigénesis ha jugado un papel protagonista. Además del material genético, el quehacer científico ha ido demostrando la importancia de los cambios epigenéticos en la diferenciación celular así como, en determinados estadios del embrión, el papel crucial de algunos metabolitos de origen materno presentes en el citoplasma de células embrionarias para su correcto desarrollo. Esta necesidad de las modificaciones epigenéticas y de lo aportado por el medio extracelular ha repercutido en el análisis de la consideración personal del embrión, y desde reflexiones filosóficas se ha discutido si los metabolitos de origen materno poseen, en el conjunto del sistema embrionario, carácter “esencial” o “constitucional” (cfr. Güell 2013).
| |
| − |
| |
| − | El objetivo de esta voz no es desarrollar estas discusiones traídas a colación. Esta breve exposición ha pretendido mostrar el gran impacto que ha supuesto la aparición de la epigenética, y cómo su irrupción, por un lado, ha hecho tambalear la comprensión de los seres vivos y su evolución y, por otro lado, ha sido fundamental para el desarrollo de estas áreas y disciplinas que suponen, en sí mismas, un terreno fértil para la reflexión interdisciplinar.
| |
| − |
| |
| − | El objetivo de esta voz es ofrecer una visión, aun general, fiel del significado de epigenética que permita comprender el desarrollo orgánico y abordar con criterio estos (y otros) debates interdisciplinares en los que se ve envuelta. Para lograr comprender su significado es necesario, primero, introducir la composición, estructura y funcionalidad del material genético para, después, analizar de modo sistemático los factores y procesos implicados en la expresión génica y en su regulación.
| |
| − |
| |
| − |
| |
| − | ==Introducción a los aspectos biológicos del material genético==
| |
| − |
| |
| − | ===Composición y estructura===
| |
| − |
| |
| − | El material genético hace referencia a los ácidos nucleicos, esto es, a las moléculas de ADN y ARN. Para los objetivos aquí propuestos introduciremos brevemente los aspectos básicos moleculares y estructurales del ácido desoxirribonucleico o ADN y del ácido ribonucleico o ARN (para profundizar en estos respectos, cfr. WATSON et al. 2008, 23-194).
| |
| − |
| |
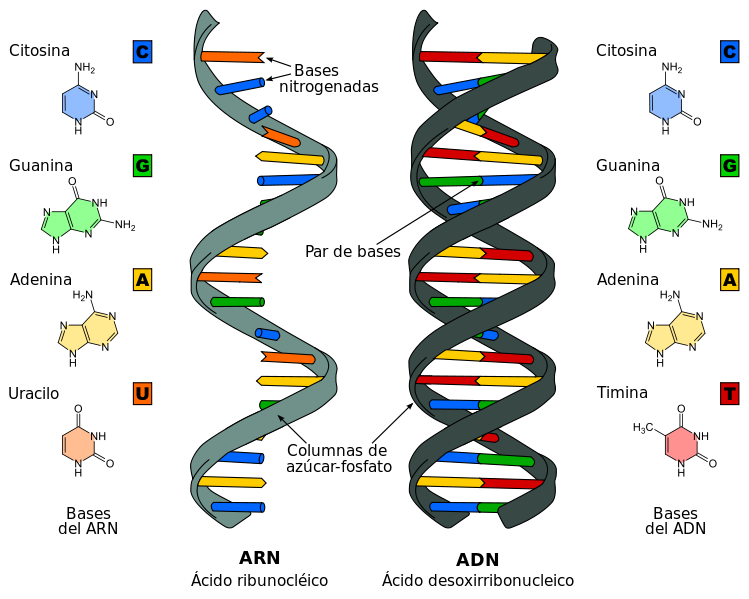
| − | El ADN es una cadena en doble hélice (véase figura 1 en Apéndice). Molecularmente es un polímero alternado de un azúcar denominado desoxirribosa y de fosfatos, y en cada monosacárido se halla unida una de las cuatro bases que componen el ADN: la adenina, guanina, timina y citosina. La unidad molecular es el nucleótido, formado por un azúcar, un fosfato y una base nitrogenada (por eso se afirma que el ADN es una cadena de polinucleótidos).
| |
| − |
| |
| − | Teniendo presente la estructura en doble hélice del ADN, mientras las columnas verticales de desoxirribosas y fosfatos son regulares, el orden de las bases (cada peldaño interno) es irregular. Esta irregularidad es la causa del contenido de lo que habitualmente se denomina “información genética” (véase la voz sobre “Información biológica” de esta enciclopedia). Más concretamente, cada peldaño está compuesto por dos bases unidas entre sí. Cada una de estas bases que forman un peldaño está inserta en una hebra, y esta unión mantiene a las hebras juntas. Las uniones entre bases están mediadas por enlaces de hidrógeno específicos: la timina de una cadena siempre se aparea con la adenina de otra, mientras que la guanina lo hace siempre con la citosina. Por ello se afirma que la doble hélice es complementaria.
| |
| − |
| |
| − | Se dice que el código genético es “universal” en el sentido de que esta molécula con esas uniones específicas (adenina-timina y guanina-citosina) está presente en todos los organismos vivos (aunque, en rigor, habría que decir “casi universal” (cfr. Watson et al. 2008, 508)). El ADN suele ser comparado con un código, receta o plano, ya que contiene las instrucciones necesarias para, con ayuda de maquinaria celular, construir otros componentes de las células (moléculas de ARN y proteínas).
| |
| − |
| |
| − | El ácido ribonucleico (ARN) conocido como ARN mensajero (ARNm) es, como el ADN, un ácido nucleico, pero está formado por una cadena lineal (y no en hélice, como el ADN) y monocatenaria (y no bicatenaria) de ribonucleótidos. Siguiendo con el símil de la escalera mencionado anteriormente para el ADN, su estructura respondería a una cadena lineal, de fosfatos y ribosas, en la que sobresaldrían las bases nitrogenadas a modo de medios peldaños. La cadena lineal respondería a una alternancia de azúcar (ribosa) y fosfato, en las que sobresaldría, unida a la ribosa, una base a modo de medios peldaños. El orden de las cuatro bases nitrogenadas posibles (en este caso, adenina, citosina, guanina y uracilo) sigue una secuencia establecida por la información presente en la molécula de ADN de la que deriva a través de un proceso de transcripción. El ARN está presente tanto en las células procariotas como en las eucariotas, y es el único material genético de ciertos virus (virus ARN).
| |
| − |
| |
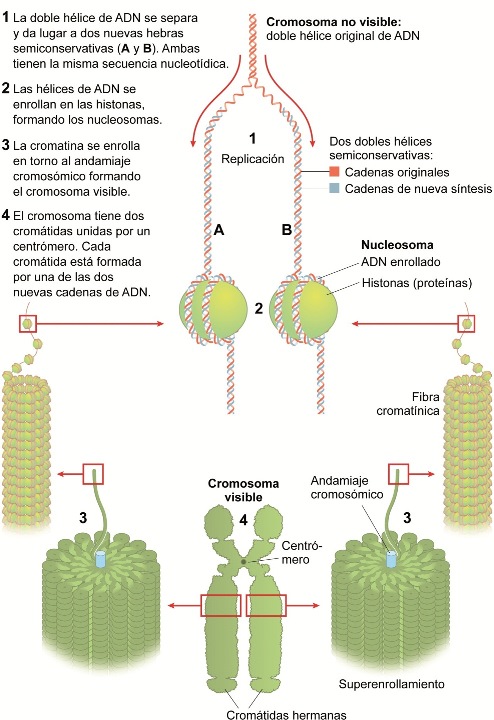
| − | Parecería que con la estructura así descrita hubiéramos agotado la estructura del material genético, pero no es así. Aunque la imagen de la doble hélice del ADN está cristalizada en el imaginario de todos, hemos de apuntar que en las células (nos centramos ahora en la de los organismos eucariotas) la doble hélice se halla compactada en una estructura filiforme denominada cromatina o fibra cromatínica. La molécula en doble hélice se encuentra rodeando unas proteínas denominadas histonas (da exactamente 1.7 vueltas en cada histona), de tal modo que una larga molécula de ADN está enrollada en una sucesión de histonas distribuidas a modo de cuentas de rosario. Al fragmento de ADN y el grupo de histonas alrededor de las que éste se enrolla se lo denomina nucleosoma. Pues bien, los nucleosomas (conectados por una misma cadena de ADN) forman hileras que, a su vez, se enrollan entre sí dando lugar a unas estructuras filiformes compactas: la cromatina o fibras cromatínicas. Esta fibra de cromatina, en determinados momentos del ciclo celular, se superenrollará a otras proteínas dando lugar a los cromosomas. Es importante tener presente que la doble hélice siempre está en las células eucariotas en forma de fibras de cromatina para, después, poder comprender la dimensión epigenética del desarrollo (véase figura 2 en Apéndice).
| |
| − |
| |
| − |
| |
| − | ===Funcionalidad===
| |
| − |
| |
| − | Aunque existe un amplio debate sobre si es o no es correcto el uso de la noción de “información” en la generalizada expresión “información genética” (para una aproximación a esta importante cuestión, cfr. Godfrey-Smith 2000 y 2007) el ADN es habitualmente presentado como aquella molécula que contiene la “información” necesaria para la consecución, a través de un largo y complejo proceso de desarrollo biológico, de un organismo. Piénsese en el cigoto, y en cómo a través de sucesivas mitosis el material genético se va replicando para que todas las células resultantes contengan toda y prácticamente la misma información genética presente desde el cigoto. Pues bien, teniendo esto presente, vamos a responder primero cómo esa información es desplegada, para, en el siguiente apartado, comprender cómo es posible que teniendo la misma información genética en todas las células de un organismo, nos encontremos con células y tejidos tan distintos.
| |
| − |
| |
| − | Por decirlo así, la información del ADN contenida en la doble hélice es “estática” y necesita ser seleccionada, copiada, filtrada, leída y traducida para que pueda ser comprendida como tal (para profundizar a este respecto, cfr. Watson et al. 2008, 369-491). A este respecto, el ARN desempeña una función crucial: mediar en el proceso de otorgar funcionalidad a la información contenida en el ADN. El proceso general puede comprenderse bien con un símil. De modo general se puede afirmar que se encuentra todo y prácticamente el mismo ADN en todas las células de un organismo. Pues bien, el ADN puede comprenderse como los tomos de una enciclopedia de recetas. Todas las células de un mismo organismo, con lo dicho, contienen los distintos volúmenes que conforman la enciclopedia, con la particularidad de que no es posible extraer ningún libro de la biblioteca en la que se encuentra, esto es, el núcleo de la célula es sólo un lugar de consulta. En la enciclopedia encontramos todas las recetas necesarias para que, siguiendo sus indicaciones y en el lugar y con los medios adecuados, se pudiera lograr diversos tipos de productos. Con lo dicho, se ha de copiar (proceso de transcripción) la receta (secuencias de ADN) de la biblioteca (núcleo). La copia de la receta ha de ser llevada a la cocina (a la proximidad de ribosomas, en el citoplasma) para que la información contenida en ella se transforme (proceso de traducción) en el producto deseado (proteínas).
| |
| − |
| |
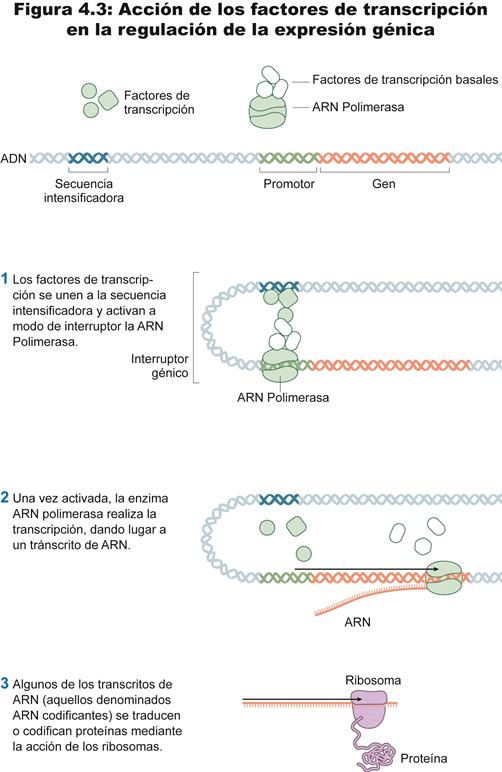
| − | A grandes rasgos (véase figura 3 en Apéndice), a través del proceso de transcripción (y por medio de la enzima catalizadora ARN polimerasa) se hace una copia de una secuencia de ADN (lo que comúnmente se ha denominado “gen”), copia que da lugar al ARN mensajero. El ARN mensajero sale del núcleo y se dirige a un orgánulo citoplasmático denominado ribosoma (en cuya estructura encontramos otro tipo de ARN, el ARN ribosómico). En asociación con el ribosoma y a través del proceso de traducción y con ayuda del ARN de transferencia, tiene lugar la síntesis de proteínas. La composición de proteínas de una célula define, en una importante medida, el tipo celular con que nos encontraremos, así que la síntesis de proteínas es la que nos indica si la célula es piel, neurona u ojo. Es interesante señalar la distinción entre ARN codificantes (los ARN mensajeros hasta ahora señalados cuyo final del camino es codificar proteínas) y los ARN no codificantes (ARNnc), esto es, secuencias de ADN cuyos transcritos no codifican proteínas (Cfr. Morris 2012). Estas moléculas de ARNnc incluyen funcionalidades abundantes y muy importantes como los ya apuntados ARN de transferencia (tRNA) y ARN ribosomal (rRNA), y otros que se encuentran actualmente bajo el punto de mira de la investigación como snoRNAs, microARNs, siRNAs y piRNAs y el ncRNA largo, con importantes funciones de regulación genética, algunas de ellas implicadas en procesos patológicos celulares (cfr. Jonas e Izaurralde 2015; Tyagi 2016).
| |
| − |
| |
| − | Antes de continuar y teniendo en cuenta lo mencionado, cabe considerar el cuestionamiento de algunos autores sobre la idoneidad de la noción de “gen”, más concretamente, del denominado ''classical molecular gene concept ''(concepto molecular clásico de gen), gen entendido como un tramo de ADN que codifica un producto funcional, ya sea una proteína o una cadena de ARN. Para comprender este cuestionamiento hay que introducir un grado más de complejidad en el proceso de expresión génica: la regulación post-transcriptacional (para profundizar, Cfr. WU 2013). Hasta ahora hemos simplificado la exposición apuntando que una secuencia de ADN daba lugar, a través del proceso de transcripción, a un ARN mensajero, y que este, tras un proceso de traducción, podía codificar a una proteína. Pues bien, el transcrito de ARN (copia de una secuencia de ADN determinada) es un “borrador” que posee unas regiones denominadas intrones que han de ser eliminadas para que el mensaje final (secuencia de regiones ensambladas denominadas “exones”) pueda ser procesado correctamente. Es como si la copia de la receta tuviera palabras de más que las hiciera incomprensibles, y hubiera que quitarlas. Entonces, es preciso apuntar que de la mera transcripción resulta una ''cadena precursora de ARN mensajero'' o ''transcrito primario'' que ha de ser procesado (cortar, eliminar y ensamblar) para originar el ARN mensajero. Y esta es, precisamente, la regulación post-transcriptacional. Pues bien, según ha podido ser comprobado, la elección de qué parte se elimina y cuál permanece de una cadena precursora de ARN o transcrito primario puede variar de tal modo que de un mismo gen (secuencia concreta de ADN) pueden llegar a producirse distintos ARN mensajeros.
| |
| − |
| |
| − | Esta regulación post-transcriptacional ha mostrado a la comunidad científica la insuficiencia de los modelos explicativos imperantes en la segunda mitad del siglo XX, que, además de no tener presente la necesaria influencia epigenética, entendían la relación entre las secuencias de ADN y sus productos como una relación de uno a uno (es decir, una secuencia concreta de ADN se transcribe en uno y siempre el mismo ARN mensajero maduro, ARN que se traduce en una y siempre la misma proteína). Desde la filosofía de la biología, algunos autores piensan que el problema ha estribado en la extrapolación gratuita de esa relación “uno a uno” de la que se tenía constancia en multitud de secuencias a todas las secuencias transcriptacionalmente activas del genoma. Teniendo esto presente, la falta de relación “uno a uno” no afecta la direccionalidad del dogma central de la biología molecular: que secuencias de ADN transcriben ARN, y que de este se sintetizan o se pueden sintetizar proteínas. En este marco parece situarse la nueva comprensión que se ofrece de la noción de gen tras los datos ofrecidos por el proyecto ENCODE como la unión de las secuencias genómicas que codifican un conjunto coherente de productos funcionales y potencialmente solapantes. Otros autores provenientes de la filosofía de la biología, por el contrario, cuestionan la utilidad del concepto de gen y proponen que la noción de “gen” y la centralidad funcional recaigan en otras estructuras, principalmente, en la unidad funcional del ARN mensajero maduro (para una introducción a la discusión véanse El-Hani y Meyer 2013 y Güell 2013, 352-353)
| |
| − |
| |
| − | Dejando a un lado esta discusión, se observa que a pesar de la centralidad del ADN, este no puede actuar solo, y el ARN gestiona esta información vital durante los procesos de transcripción y traducción, además de participar en actividades catalíticas y de regulación génica.
| |
| − |
| |
| − |
| |
| − | ===El silenciamiento de genes===
| |
| − |
| |
| − | La influencia epigenética apunta a los factores involucrados en la regulación de la expresión génica de células (y tejidos) en cada etapa específica del desarrollo. La expresión génica es el proceso por el cual la información de la doble hélice de ADN se convierte, por el proceso de transcripción, en transcritos de ARN, y este, mediante el proceso de traducción, en proteínas. La transcripción es pues el primer paso de la expresión génica, y comprende la copia de secuencias de ADN, los comúnmente denominados genes, en secuencias de ARN, el denominado ARN mensajero. Este proceso es catalizado por la enzima ARN polimerasa. Solo algunas regiones del genoma se transcriben, y las regiones elegidas varían en células diferentes o en diversos momentos del desarrollo de cada una de ellas. Las distintas regiones pueden transcribirse en grado diverso, una sola o miles de veces. El segundo paso de la expresión génica es la traducción. La traducción comprende la decodificación de la información de secuencias de nucleótidos que configura el ARN mensajero en las proteínas (más concretamente, en la secuencia lineal de los aminoácidos que configura una cadena polipeptídica, cadena constitutiva de las proteínas). La trascripción se produce en el núcleo, pero la traducción tiene lugar en el citoplasma (en los ribosomas).
| |
| − |
| |
| − | Si todas las células de un organismo poseen el mismo material genético, ¿por qué las células y los tejidos son tan distintos? La clave está en percatarse de que aunque tenemos en todas las células toda la información para dar lugar a cualquier tipo de célula, cada célula está tan sólo transcribiendo y traduciendo las proteínas que le son propias y la caracterizan. Las células del músculo, aun teniendo la información para originar proteínas características de las neuronas, poseen un fenotipo muscular ¿Por qué? ¿Cómo ocurre esto? Intuitivamente la respuesta es sencilla: aunque en el ADN encontramos todos los tomos, aquellos que no correspondan al tejido celular en cuestión estarán inaccesibles para ser transcritos. De la comprensión sobre los mecanismos que permiten abrir y cerrar el acceso a determinadas regiones del genoma va a depender la correcta comprensión de la epigenética, y para ello, se ha de profundizar más en los factores que intervienen en la transcripción.
| |
| − |
| |
| − | Como fue mencionado anteriormente, la ARN polimerasa es la enzima encargada de llevar a cabo la transcripción, esto es, la síntesis de un transcrito primario de ARN a partir de una secuencia del ADN. Solo algunas regiones del genoma se transcriben, y las regiones elegidas varían en células diferentes o en diversos momentos del desarrollo de cada una. Además, las distintas regiones pueden transcribirse en grado diverso, desde una sola vez a miles de veces, y todo ello dependerá, principalmente, de la actividad de la ARN polimerasa y otros factores de transcripción asociados.
| |
| − |
| |
| − | La ARN polimerasa se une a la doble hélice para llevar a cabo una copia a partir de una secuencia concreta de ADN. Para ello, se ancla en una región adyacente a un gen para, después, comenzar un “barrido” por ese gen que tiene como resultado la síntesis de una copia en ARN a partir de la secuencia de ADN en cuestión. Esa región de ADN donde se ancla la ARN polimerasa se llama “promotor”, y tiene una relevancia muy especial: puede estar “obstruida” no permitiendo el anclaje de la ARN polimerasa, lo que da lugar a un gen silenciado. He aquí el mecanismo para poner bajo llave determinados genes o regiones del ADN: obstruir el acceso de la ARN polimerasa (o, de modo más general, a los factores de transcripción). Hay dos modos distintos de “obstruir” regiones en el genoma y, por ende, de silenciar secuencias donde la transcripción es posible (es decir, de inactivar genes). Para explicarlos, ha de traerse de nuevo a colación la estructura del ADN.
| |
| − |
| |
| − |
| |
| − | ==Regulación epigenética==
| |
| − |
| |
| − | ===A nivel de citosinas===
| |
| − |
| |
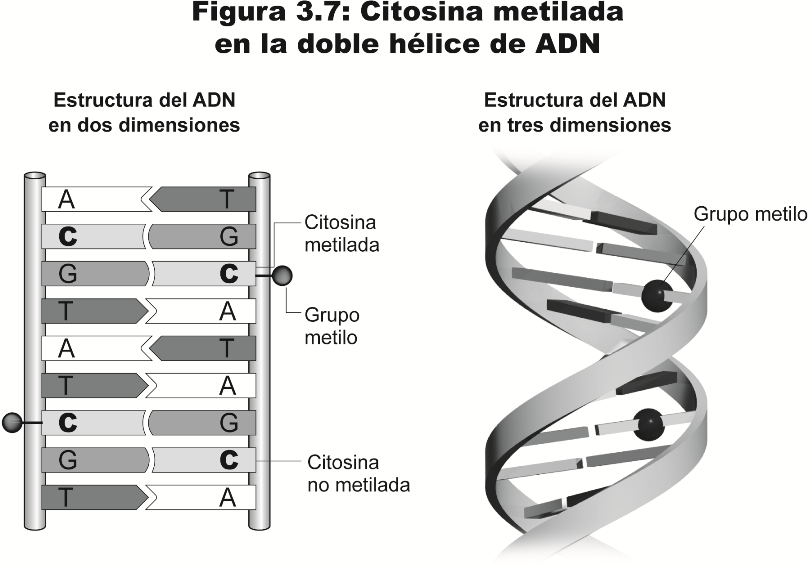
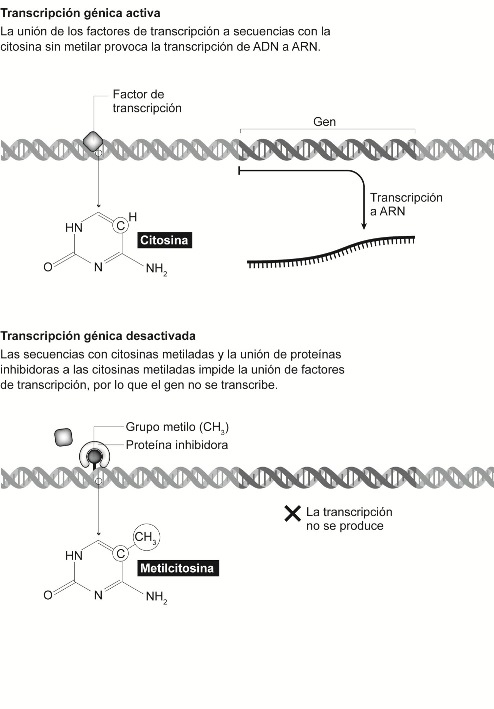
| − | Uno de los modos de “obstruir” la acción de la ARN polimerasa responde a un proceso químico: '''la metilación de las citosinas '''(véase figura 4 en Apéndice) (Jones y Takai 2001). Recordamos ahora que el ADN está formado por una cadena en doble hélice donde, en el interior de la hélice, encontrábamos la conjunción de dos bases nitrogenadas, que podrían ser guanina-citosina o adenina-timina. Para su actividad, la ARN polimerasa se une a unas regiones de la secuencia de ADN adyacentes a un gen denominadas promotores, regiones que tienen la particularidad de contener una gran cantidad de citosinas (más concretamente, gran densidad de regiones denominadas islas CpG, donde la “p” hace referencia al fosfato que une ambos nucleótidos). En el ADN de vertebrados se metila usualmente el carbono 5 de las citosinas, y la metilación de determinadas citosinas inhibe la unión de factores de transcripción e inactiva al gen imposibilitando la expresión génica. La marca química es, por decirlo así, como las agujas que encontramos en las repisas de algunos monumentos históricos para que las palomas no se posen.
| |
| − |
| |
| − | Además de esto, encontramos que algunas secuencias de ADN solo son reconocidas por represores específicos de la transcripción cuando están metiladas. Veremos a continuación que estos represores que se unen a las citosinas metiladas desactivan los genes cercanos mediante el reclutamiento de histonas desacetilasas. Es suficiente por ahora comprender que las agujas, además de obstruir por sí mismas el anclaje de la maquinaria de transcripción, pueden ser reconocidas por moléculas que, uniéndose a ellas (a las agujas), hagan más eficaz su cometido y, a su vez, dificulten la reversibilidad de la secuencia a un estado de transcripción posible (véase figura 5 en Apéndice)
| |
| − |
| |
| − | Por lo tanto, el patrón de metilación del ADN de una célula hace referencia al grado de metilación que encontramos en aquel genoma. Este patrón es una información interesante, pero demasiado global: nos ofrece una idea de cómo de metilado está el ADN de una célula, pero no nos indica qué regiones del genoma están metiladas. Con el paso de los años se ha ido afinando en ir reconociendo qué lugares de qué tejidos están metilados, lo que ha sido el objeto de estudio de proyectos de investigación globales como el IHEC y el ENCODE antes mencionados.
| |
| − |
| |
| − |
| |
| − | ===A nivel de histonas===
| |
| − |
| |
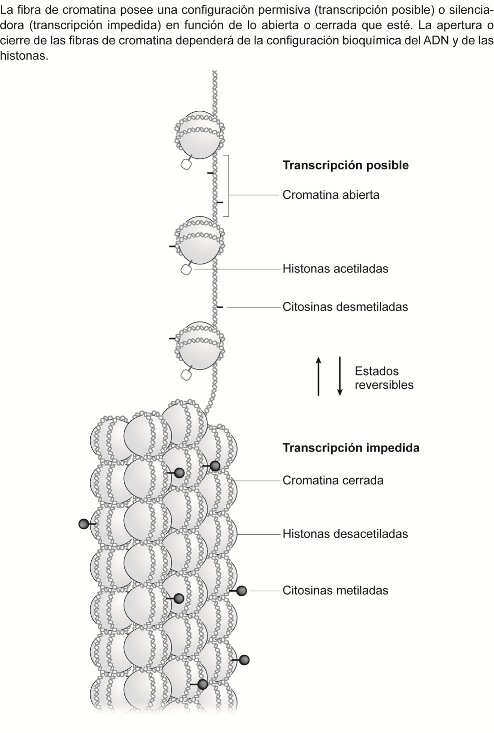
| − | Otro de los modos de obstruir el acceso a la maquinaria de transcripción es a través de '''''modificaciones químicas a nivel de histonas''''' (véase figura 6 en Apéndice). Determinadas modificaciones químicas en las histonas alteran la configuración espacial de la fibra cromatínica produciendo una compactación de los nucleosomas que impide el acceso de la maquinaria de la transcripción y, por ende, inactiva el gen en cuestión.
| |
| − |
| |
| − | Hay que tener presente que la actividad transcriptacional basal es esencialmente restrictiva: los promotores están en estado inactivo hasta que se ponen en marcha por la acción de los elementos llamados “activadores” y el ensamblaje de la maquinaria de transcripción. Para que comience la transcripción del ADN es necesario, entonces, que la cromatina de esa región esté relativamente descondensada para que quede expuesta más fácilmente la doble hélice y se permita el acceso de la maquinaria necesaria para que se inicie la transcripción.
| |
| − |
| |
| − | Los nucleosomas son, por tanto, un elemento fundamental para mantener este estado basal restrictivo, pues impiden el acceso de la maquinaria transcriptacional a los promotores inhibiendo entonces la expresión génica. Pues bien, como se ha apuntado, determinadas proteínas tienen la capacidad de, uniéndose a la estructura del ADN metilado, producir plegamientos que activan o silencian determinados genes. Estas proteínas pueden ser nucleosómicas (es decir, histonas modificadas) o no nucleosómicas. Las mejor conocidas son las nucleosómicas. Los nucleosomas están compuestos por un octámero de cuatro histonas. Sus colas N-terminal son objeto de más de 100 modificaciones diferentes post-translacionales, incluyendo acetilación, metilación, fosforilación y ubiquitinación, ya sea para activar o inhibir los efectos de transcripción. Por ejemplo, se conoce que la acetilación de las histonas conduce a la activación transcripcional, y la desacetilación de las histonas crea una estructura cromatínica más condensada que favorece el silenciamiento de la transcripción de los genes incluidos en dicha región genómica.
| |
| − |
| |
| − | Asimismo, ha sido descrito el silenciamiento temporal de genes por medio de histonas modificadas en genes que tienen que ser silenciados en las primeras etapas del desarrollo, para posteriormente ser activados. La modificación de histonas es un mecanismo fácilmente reversible cuando la expresión del gen silenciado es necesaria.
| |
| − |
| |
| − |
| |
| − | ===Caracterización de las marcas epigenéticas===
| |
| − |
| |
| − | Es importante apuntar al carácter '''hereditario''' que pueden tener las marcas epigenéticas. No sólo el ADN se hereda generacionalmente entre individuos y, a través de la mitosis, entre generaciones de células. Las marcas epigenéticas también pueden ser heredadas, jugando un papel esencial tanto para la consecución de la reproducción entre individuos como para una adecuada diferenciación celular.
| |
| − |
| |
| − | En lo que respecta a la reproducción, se dice que los gametos masculinos y femeninos han de estar maduros. Esta maduración (que el organismo regula hormonalmente) se traduce en una configuración bioquímica y espacial del ADN –o lo que es lo mismo, en una configuración epigenética- asimétrica concreta no solo necesaria para que se logre la fecundación, sino para que el desarrollo del individuo sea adecuado. Que la configuración sea '''asimétrica''' significa que el grado de metilación del material genético de una célula posee una dinámica de transformación distinta, desde el inicio de la fecundación y a lo largo del desarrollo, según si su origen es materno o paterno: esto, a su vez, se traduce en que el nivel de expresión de los genes cambia según si los mismos fueron heredados de la madre o del padre.
| |
| − |
| |
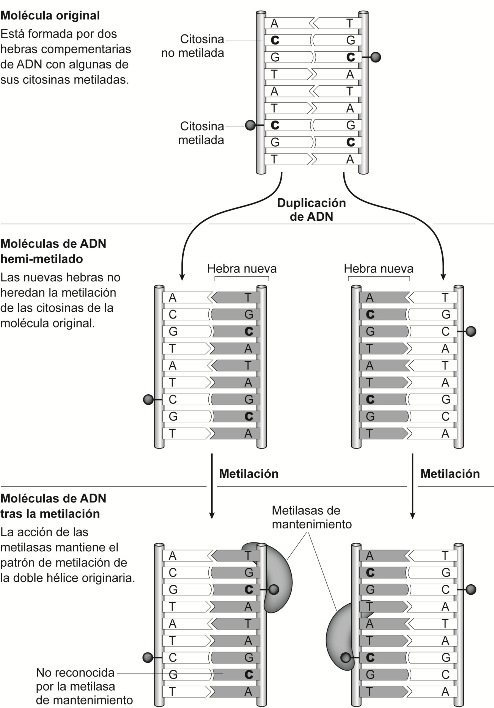
| − | Si queremos entender la herencia epigenética que se da entre generaciones de células hay que percatarse de que la mitosis tiene carácter semiconservativo (véase figura 2 en Apéndice).''' '''Cuando una célula, por mitosis, origina dos nuevas células, el material genético de las nuevas células ha heredado tan sólo una hebra de la doble hélice originaria, y la otra hebra es, necesariamente, de nueva síntesis. Hay, por lo tanto, una herencia de la mitad de las marcas existentes en la doble hélice original, y para mantener el mismo perfil de expresión génica en las nuevas células, habrá que volver a marcar químicamente aquellos lugares donde la marca se perdió. A este respecto, se han descubierto enzimas capaces de metilar esas regiones (las metilasas de mantenimiento, véase figura 7 en Apéndice), pero también es posible reprogramar determinadas regiones a través de la metilación de nuevas regiones (metilasas de novo), o desmetilar regiones (desmetilasas), modificando así la expresión génica de la nueva generación de células y, por ende, originando células y tejidos diferentes. Se entiende así la dinámica de la diferenciación celular: con el transcurso del desarrollo orgánico desde el cigoto, la configuración epigenética de las células va especializándose y comienzan a aparecer nuevos grupos de células que forman capas y tejidos diferenciados. He aquí el carácter '''diferencial '''de la dimensión epigenética, responsable de la diferenciación celular. Pero, además, se entiende también el carácter '''reversible''': la acción de metilasas y desmetilasas permite reprogramar las células a distintos estadios del desarrollo, posibilitando, como después veremos, las inducción de células madre pluripotentes (las células madre pluripotentes inducidas o iPSCs) o la clonación por transferencia nuclear. Se entiende ahora, por todo lo dicho, que las marcas epigenéticas poseen un carácter '''dinámico'''. La dimensión epigenética es dinámica porque la configuración bioquímica y espacial del ADN va modificándose desde la interacción de los gametos a lo largo del desarrollo gradual y asimétricamente entre las distintas células y tejidos del organismo. En las células somáticas del organismo adulto, en las que cabría esperar un grado de metilación estable, estudios han demostrado que este se va modificando conforme avanza la edad del organismo adulto, y que, además, los cambios son similares en individuos de la misma familia (Bjornsson et al. 2008).
| |
| − |
| |
| − | Teniendo presente todas estas características, es más fácil de comprender que la alimentación, el estilo de vida y factores medioambientales pueden exponer al organismo a situaciones que generan respuestas químicas capaces de producir cambios en el epigenoma, algunos de los cuales pueden ser perjudiciales. He aquí el potencial patológico de la dimensión epigenética que será retomada en el último apartado.
| |
| − |
| |
| − |
| |
| − | ==Aclaraciones terminológicas==
| |
| − |
| |
| − | ===Influencia epigenética y marcas epigenéticas===
| |
| − |
| |
| − | Habitualmente, la literatura científica y de corte interdisciplinar entiende como mecanismos epigenéticos a aquellos factores involucrados en la regulación de la expresión génica de células (y tejidos) en cada etapa específica del desarrollo. Dentro de este amplio campo, están implicadas la metilación del ADN, la modificación de las histonas, las variantes de las histonas y el posicionamiento de nucleosomas, así como los ARN no codificantes y la maquinaria envuelta en los procesos de transcripción. Y todos lo están por influir en la estructura de la cromatina y la modulación de las interacciones entre las proteínas y el ADN que, como hemos apuntado, regulan la expresión génica. Desde esta perspectiva, por ejemplo, la influencia de las hormonas maternas recibidas sobre el embrión durante la implantación, del humo del tabaco en las células pulmonares, de los rayos de sol en la piel o del estrés sufrido son también “factores epigenéticos” capaces de originar una cascada de mecanismos que alterarán la epigenética. Desde esta perspectiva se puede hablar de “influencia epigenética”.
| |
| − |
| |
| − | Si en vez de apuntar a “mecanismos” epigenéticos, nos referimos a las “marcas” epigenéticas, el área comienza a delimitarse, pues las marcas hacen referencia no a la cascada de reacciones celulares, sino al último eslabón de la cadena: a las modificaciones químicas del ADN, o de proteínas asociadas a él, que, sin alterar la secuencia de nucleótidos, influyen en la funcionalidad del mismo activando o silenciando determinados genes.
| |
| − |
| |
| − | Haciendo la distinción entre “mecanismos epigenéticos” y “marcas epigenéticas”, es más fácil percatarse de que siempre nos encontramos con un “punto de partida” no sólo genético, sino también epigenético; es decir, con una configuración concreta intrínseca de la que parte toda célula del organismo desde su estadio unicelular y que, además de poder ser afectada por el ambiente, va siendo afectada y necesitada de las influencias del medio externo para su correcto dinamismo. Tener esta distinción presente permite abordar la influencia del ambiente en el desarrollo orgánico sin olvidar que las marcas epigenéticas de una célula también dependen directamente de la configuración de la secuencia genómica (y de los metabolitos existentes en el citoplasma) de la célula de la que, por mitosis, proviene. Se puede hablar entonces de una comprensión de la epigenética pro-activa o reactiva. La pro-activa está relacionada directamente con la herencia (se ve en el compromiso de diferenciación paulatino del desarrollo). La comprensión re-activa presupone una sensibilidad de la función génica al ambiente externo donde mecanismos epigenéticos originados en el exterior de organismos pueden modificar la expresión génica dando lugar, con una misma secuencia genómica, a fenotipos variables (Bird 2007).
| |
| − |
| |
| − |
| |
| − | ===Genoma===
| |
| − |
| |
| − | La palabra '''genoma''' hace referencia al conjunto de la información genética de una célula, siendo paradigmático del término todo el conjunto del ADN contenido en el juego diploide presente en el cigoto, y, a partir del cigoto, en el resto de células que componen un organismo eucariota adulto. En el caso del hombre corresponde a aproximadamente 3200 millones de pares de nucleótidos, los cuales, replicados y superenrollados, nos aparecen de forma visible y nítida en 23 pares de cromosomas en la metafase (aunque no es relevante para el análisis aquí presente, cabe señalar que el interior de las mitocondrias contiene moléculas circulares de ADN de 16.559 pb que albergan 37 genes y se transmiten por vía materna) (cfr. Jouve 2008, 478).
| |
| − |
| |
| − | Como es sabido, en el genoma están contenidos los patrones que posibilitarán la transcripción de estas secuencias a ARN y la traducción de este a proteínas. De los 3200 millones de pares de bases del genoma humano, se ha afirmado que unos 1200 millones corresponden a los genes (un total estimado de 25000) y a secuencias relacionadas con ellos, representando el 36 por cien del total de genoma. Cabe apuntar que de ese 36 por cien, tan solo el 1,5 contiene información que se traducirá a proteínas, o lo que es lo mismo: del cien por cien del genoma tan solo el 1,5 por cien son secuencias con información de genes estructurales. El 62,5 por cien del genoma que no está relacionado directamente con los genes los 2000 millones de pares de bases restantes es denominado “gen intragenético”, siendo algo menos de las tres cuartas parte del mismo secuencias repetidas (Cfr. Watson 2008, 149, fig. 7-4).
| |
| − |
| |
| − | Estas cifras proporcionadas principalmente por el “Proyecto Genoma Humano” han sido completadas y corregidas con otro proyecto del ''National Human Genome Research Institute'' (NHGRI), el proyecto ENCODE (acrónimo de ''Encyclopedia of DNA Elements''). El ENCODE comenzó como proyecto piloto en el 2003, y durante la última década se ha encargado de hacer un análisis exhaustivo del genoma humano, centrándose en identificar los elementos funcionales de la secuencia de ADN humano que nos encontramos en las distintas líneas celulares. En los últimos años se ha ampliado de forma significativa la información sobre los transcritos primarios y maduros, así como la localización de las principales modificaciones de histonas, los sitios de unión de los factores de transcripción, los sitios de inicio de la transcripción, etc. y todo ello unido a datos de expresión génica, de replicación y del número de copia de esas mismas regiones. (cfr. Nature ENCODE Explorer).
| |
| − |
| |
| − | Merece la pena detenernos en algunos de los sorprendentes resultados de este proyecto. Se ha visto que aproximadamente el 90 por cien de los nucleótidos del genoma tienen alguna función en sentido amplio, es decir, son transcritos en al menos un tipo celular. Se ha visto también que hay más de ocho millones de sitios en el genoma a los que se une algún factor de transcripción, lo cual ha permitido entender mucho mejor la especificidad de la expresión génica del tejido. Además, según sus resultados, parece claro que una región genómica puede codificar distintos productos proteicos y además dar lugar a otros transcritos (codificantes o no codificantes) en ambas cadenas. Esto nos da una idea de la complejidad de la dinámica funcional genética, y de cómo la ciencia está avanzando a este respecto y abriendo nuevos horizontes (Novo 2016).
| |
| − |
| |
| − |
| |
| − | ===Epigenoma===
| |
| − |
| |
| − | Introducir la noción de '''epigenoma '''ayuda a comprender adecuadamente la relación entre el ADN y el resto de estructuras. Encontramos en la literatura académica que este término suele ser entendido como la suma de las marcas químicas del ADN o la cromatina “que están literalmente encima del genoma” (''Oxford Dictionary of Biology'' 2015, 204) cuya modificación no implica el cambio de secuencias de nucleótidos. En el marco del ya anunciado Consorcio Internacional del Epigenoma Humano, con el término epigenoma se hace referencia a la realización de un mapa genómico que abarque las modificaciones epigenéticas, es decir, la confección de un mapa que recoja no solo la secuencia nucleotídica (lo que viene llevando a cabo el Proyecto Genoma Humano) sino que plasme la influencia epigenética y la regulación de esta en la expresión génica. Estas marcas hacen referencia principalmente a la metilación del ADN en los nucleótidos con citosina, la modificación de histonas y sus variantes incorporadas a los nucleosomas (cfr. Egli, Brkhoff y Eggan 2008, Box 1). Otra definición del término no se enfoca ni en las marcas químicas ni a un mapa, sino en la estructura resultante, es decir, en la configuración bioquímica del ADN y la configuración bioquímica y estructural de las fibras de la cromatina que la célula posee en un determinado estadio del desarrollo celular (cfr. Güell 2013). Esta tercera vía entiende que la presencia física del genoma (secuencia polinucleotídica) no se puede entender sin una precisa configuración estructural y bioquímica. Por este motivo, aludir a la configuración bioquímica y espacial con ese término ofrece una comprensión levemente distinta a la de mapa en abstracto o al epigenoma como información independiente que posee el ADN a modo de marcas “encima del genoma”. Esta tercera vía se centra el estado físico concreto de cada cadena desoxirribonucleica imbricada indisolublemente en una estructura cromatínica espacialmente situada, estado, ciertamente, causado por la ubicación de las marcas epigenéticas e implicado inapelablemente en la dinámica de proliferación y diferenciación celular con un metabolismo definido.
| |
| − |
| |
| − | Por otro lado, se mencionó anteriormente que las modificaciones epigenéticas hacen referencia en la mayoría de los casos a la unión reversible de ciertos grupos químicos al ADN y a las fibras cromatínicas lo que da como resultado una alteración de la capacidad de transcripción (sin que ello implique un cambio en la secuencia del genoma). Se decía también que en la regulación de estos grupos químicos pueden estar implicados metabolitos citoplasmáticos sintetizados por la propia célula, provenientes de células vecinas o incorporados del exterior del organismo. Aunque sólo estos últimos son los usualmente denominados ambientales, el “'''ambioma'''” también incluye la influencia de origen en el propio organismo (por ejemplo, las hormonas provenientes de tejidos distantes a través de la circulación sanguínea).
| |
| − |
| |
| − | La regulación génica, según lo dicho, recibe influencia del ambioma concreto en el que se encuentre la célula o el organismo en cuestión (Jaenisch y Bird 2003). Pongamos un ejemplo de amplio alcance. En el ciclo menstrual las hormonas inducen el desarrollo de un folículo en cuyo interior va a madurar un ovocito que resultará, en la ovulación, un óvulo maduro. Se dice que el óvulo está maduro cuando el ADN y las fibras cromatínicas han alcanzado una configuración concreta capaz, por un lado, de sintetizar todos los productos necesarios para que, una vez fecundado, el organismo tenga todo lo necesario para su correcto desarrollo; y, por otro lado, para que en el óvulo, una vez fecundado, el ADN de origen materno entre en un funcionamiento sincrónico adecuado con el ADN de origen paterno. Como se explicó más arriba, en el cigoto y en todas las células de los organismos derivadas de este los juegos de ADN paterno y materno poseen una configuración asimétrica (es decir, una expresión génica distinta) pero sincronizada. En este ejemplo, las hormonas forman parte del ambioma en lo que respecta a la dinámica epigenómica de los gametos.
| |
| − |
| |
| − | Con lo hasta aquí explicado, en el epigenoma de una célula están presentes la secuencia nucleotídica (el genoma) con su configuración bioquímica y espacial concreta en constante cambio por los efectos de su propio metabolismo y del ambioma, del que recibe influencias de células vecinas o lejanas, o del medio ambiente externo al organismo. Por último, la realidad física y conductual resultante del despliegue de la realidad biológica imbricada indefectiblemente en un ambiente es lo que denominamos '''fenotipo'''.
| |
| − |
| |
| − |
| |
| − | ==La dimensión epigenética el desarrollo==
| |
| − |
| |
| − | El genoma es la colección completa de ADN de un organismo que hace de cada individuo algo único. La secuencia de nucleótidos de ADN contiene un código que, una vez expresado (y tras una regulación post-transcriptacional) codifica los productos funcionales (ARN no codificantes y proteínas) que, junto con el resto de estructuras celulares, llevan a cabo una variedad de funciones celulares. El ADN y las proteínas asociadas poseen unas marcas químicas que confieren a las fibras cromatínicas una configuración bioquímica y espacial, el epigenoma, responsable de la activación o desactivación de genes y, por ende, del control de la funcionalidad en células específicas sin modificar la secuencia del ADN.
| |
| − |
| |
| − | La diferenciación celular y, consecuentemente, el desarrollo orgánico es fruto de la progresiva, ordenada y coordinada expresión de la información contenida en el epigenoma, expresión regulada por y necesitada de cambios epigenéticos. Estos cambios epigenéticos están regulados por la propia dinámica funcional del ADN y por procesos y metabolitos celulares cuyo origen puede ser celular o proveniente del medio.
| |
| − |
| |
| − | Dicho esto podemos afirmar que factores epigenéticos intracelulares y ambientales están directamente implicados, a través de reacciones en cadena, en el cómo y cuándo de la transcripción y traducción del ADN mediante mecanismos de activación y silenciamiento de secuencias concretas, motivo por el cual poseen una relevancia en el desarrollo orgánico y, por ende, influencia en la salud y el comportamiento. Algunos ejemplos nos ayudarán a comprender la relevancia de la dimensión epigenética.
| |
| − |
| |
| − | Con respecto a la '''diferenciación tisular''', encontramos que, en un organismo, las células de la retina poseen la misma secuencia de ADN (lo que se denomina usualmente como “genoma”) que las células de la piel gracias al proceso de replicación culminado en sucesivas mitosis desde el cigoto; pero, evidentemente, el epigenoma y la composición celular global de ambas es distinta. Las diferencias epigenéticas son responsables de que el apogenoma (el conjunto de genes expresados de una célula) entre una y otra difieran. Ello hace que el transcriptoma y el proteoma den como resultado junto con otros factores tales como la posición de cada célula dentro del conjunto un fenotipo distinto. En el desarrollo del embrión preimplantatorio se observa lo mismo: aunque el epigenoma y, consecuentemente, el apogenoma del embrión en su estadio de dos blastómeros es ya distinto en cada célula, se ve con más claridad cuando en el estadio de 8 blastómeros unas células se diferenciarán en trofoblasto y otras en embrioblasto, cada una con fenotipo distinto.
| |
| − |
| |
| − | La gemelación es, también desde el punto de vista epigenético, un acontecimiento de enorme interés. En las parejas de '''gemelos monocigóticos''', a pesar de que comparten un genotipo común, se pueden observar varios tipos de discordancia fenotípica tales como las diferencias en la susceptibilidad a las enfermedades y una amplia gama de características antropomórficas. Hay varias explicaciones posibles para estas observaciones, pero una es la existencia de diferencias epigenéticas. Para abordar esta cuestión, un estudio examinó las diferencias globales y específicas en la metilación del ADN y la acetilación de histonas de una gran cohorte de gemelos monocigóticos. La investigación encontró que aunque los gemelos son indistinguibles epigenéticamente durante los primeros años de vida, en edad avanzada los gemelos monocigóticos mostraron diferencias notables (Fraga et al. 2005)
| |
| − |
| |
| − | La generación de organismos clónicos a través de '''transferencia nuclear''' ofrece una visión de conjunto adecuada sobre el papel de la influencia epigenética. En este subapartado expondremos el paradigmático caso de la oveja Dolly, primer mamífero clónico (Griffin 1999) logrado por Ian Wilmunt (Campbell et al. 1996). El equipo de Wilmunt cultivó células epiteliales de oveja de 6 años de la raza Finn Dorset preñada. Tras obtener oocitos de distintas razas de ovejas, procedieron a introducir el núcleo de las células epiteliales en los oocitos enucleados. La fusión del núcleo al oocito la lograron con pulsos eléctricos en ambas membranas gaméticas, pulsos que estimulaban a su vez al cigoto a comenzar el desarrollo. De 430 oocitos fertilizados, se obtuvieron 277 sistemas unicelulares, los cuales se cultivaron por separado durante 6 días. Tan solo se obtuvieron 29 blastocistos aparentemente normales, los cuales se transfirieron a 29 hembras reproductoras. El único éxito fue un embrión implantado en una hembra Scottish Blackface, cuyo resultado fue la mediática oveja Dolly. Los otro 28 murieron en estadio de fetos o neonatos con alteraciones en el desarrollo. Lo que ocurre en la clonación no es difícil de entender después de lo expuesto hasta ahora. Se produce un borrado de las marcas epigenéticas propias del tejido epitelial del genoma de la célula adulta transferida y se establece un epigenoma de carácter totipotencial.
| |
| − |
| |
| − | Otro hecho que muestra con claridad la influencia de la epigénesis es lo que acontece en las '''abejas'''. Se ha descubierto que las abejas reinas y sus trabajadoras son clones. A pesar de la idéntica secuencia de ADN (mismo genoma), las reinas y sus trabajadores tienen diferentes comportamientos, morfologías y capacidad reproductiva. La diferenciación estriba en que las reinas han sido aquellas larvas, a diferencia del resto, alimentadas exclusivamente con jalea real. La composición de la jalea real causante de tal efecto se desconoce, pero sus efectos en gran medida se pueden imitar al disminuir los niveles de un solo regulador epigenético, la ADN metiltransferasa DNMT3 (Kucharski et al. 2008). En ese caso, la influencia epigenética aportada desde el ambioma en el caso del huevo destinado a ser reina es determinante para que, aun con la misma secuencia que sus clones, su fenotipo sea profundamente distinto a todos ellos. Y lo es porque los epigenomas resultantes de los tejidos de la reina difieren de los epigenomas de sus clones trabajadores.
| |
| − |
| |
| − | La influencia y fuerza de la epigénesis en el desarrollo de individuos con una misma secuencia de ADN queda patente en el ámbito de la gemelación y de la clonación, y muestra la necesidad de una descripción del hecho biológico que se aleje de lo estático (genoma) para captar la complejidad del dinamismo del desarrollo orgánico (epigenoma).
| |
| − |
| |
| − | Otro ejemplo que ayuda a entender la dimensión epigenética es el descubrimiento de las '''células madre pluripotenciales inducidas (iPSCs)'''. La reprogramación de un núcleo ya diferenciado en totipotencial supuso la puesta en práctica de las técnicas de clonación y animó al premio nobel Shinya Yamanaka a indagar en los mecanismos con un objetivo preciso: lograr deshacer el camino recorrido por las células especializadas e inducirlas a estados de indiferenciación. La inducción a la pluripotencialidad de células somáticas a través de la incorporación de, en un primer momento, genes utilizando vectores víricos (Takahashi y Yamanaka 2006) y, en un segundo momento, mediante la introducción de vectores multi-proteicos (Stadtfeld et al. 2008), permitieron la obtención de las iPSCs. Su existencia es prueba indiscutible de la importancia de los genes en el destino y diferenciación de las diferentes líneas celulares embrionarias y, a su vez, de la importancia de los factores epigenéticos en el devenir del sistema (Kim et al. 2010).
| |
| − |
| |
| − | Teniendo presente lo que acontece en las abejas, y la caracterización de las marcas epigenéticas como heredables, reversibles, asimétricas y diferenciales, es más fácil de comprender que la alimentación, el estilo de vida y los factores medioambientales pueden exponer al organismo a situaciones que generan respuestas químicas susceptibles de producir también cambios en el epigenoma, algunos de los cuales pueden ser perjudiciales. He aquí el potencial patológico de la dimensión epigenética. El '''cáncer''' es uno de los campos más estudiados a nivel molecular a este respecto (Johnson et al. 2015). En la Red del ''The Cancer Genome Atlas'' (NIH 2016), por ejemplo, se están comparando cambios en la secuencia del ADN y cambios en el número de grupos metílicos en el ADN entre células normales y tumorales. Actualmente hay muchas líneas de investigación que tratan de comprender el efecto del ambiente a través de las modificaciones epigenéticas en las enfermedades y los mecanismos a través de los cuales estas podrían ser eventualmente heredables por varias generaciones. Encontramos estudios que muestran cómo '''episodios traumáticos y de estrés''' en la infancia vienen acompañados por modificaciones epigenéticas siendo estas vivencias uno de los principales factores para el desarrollo de enfermedades mentales en el adulto (Mitchell, Schneper y Notterman 2016). Aunque encontramos trabajos que apuntan a una herencia epigenética transgeneracional (Babenkoa et al. 2015), otros estudios sostienen que no existe aún evidencia conclusiva sobre dichos efectos en la salud de los humanos. (Martos Tang y Wang 2015).
| |
| − |
| |
| − | Otro de los campos del ámbito científico en plena ebullición que ayuda a comprender el efecto del ambiente en la dimensión epigenética del desarrollo es el estudio del impacto de los procedimientos utilizados en las técnicas de '''Reproducción Humana Asistida''' (RHA) en la salud del niño nacido. La maduración de los gametos y el desarrollo del embrión temprano son las etapas más vulnerables del desarrollo, ya que las modificaciones epigenéticas son esenciales para el adecuado desarrollo del organismo. Ya hemos explicado la centralidad de la dinámica epigenética para la maduración de los gametos, y los avances en la investigación en este campo muestran el impacto de los procesos técnicos involucrados en la RHA en la dinámica epigenética y celular que, en la práctica, se traduce en la escasa eficacia de la técnica (Tarín et al. 2014) y en el aumento de patologías de la población en cuestión (Gutarra-Vilchez et al. 2013; Scherrer et al. 2015) cuyo vínculo epigenético es evidente (Padhee et al. 2015).
| |
| − |
| |
| − |
| |
| − | ==Bibliografía==
| |
| − |
| |
| − | Avery, Oswald T.; MacLeod, Colin M. y McCarty, Maclyn. 1944. “Studies on the Chemical Nature of the Substance Inducing Transformation of Pneumococcal Types - Induction of Transformation by a Desoxyribonucleic Acid Fraction Isolated from Pneumococcus Type III”. ''Journal of Experimental Medicine'' 79 (2):137–158.
| |
| − |
| |
| − | Dickens, T. y Rahman, Q. 2012. “The extended evolutionary synthesis and the role of soft inheritance in evolution”. ''Proceedings of the Royal Society: B biological sciences'' 279 (1740): 2913-2921.
| |
| − |
| |
| − | Babenkoa, O., Kovalchukb, I. y Metz, G.A.S. 2015. “Stress-induced perinatal and transgenerational epigenetic programming of brain development and mental Health”. ''Neuroscience & Biobehavioral Reviews'' 48:70–91
| |
| − |
| |
| − | Bird, A. 2007. “Perceptions of epigenetics”. ''Nature'' 447: 396–398.
| |
| − |
| |
| − | Campbell, K.H., Mcwhir, J., Ritchie, W.A. y Wilmut, I. 1996. “Sheep cloned by nuclear transfer from a cultured cell line”. ''Nature ''380: 64-66.
| |
| − |
| |
| − | Editorial. 2010. “Time for epigenome”. ''Nature'' 463:587.
| |
| − |
| |
| − | Egli, D., Brkhoff, G. y Eggan, K. 2008. “Mediators of reprogramming: transcription factors and transitions through mitosis”. ''Molecular Cell Biology'' 9:506.
| |
| − |
| |
| − | Fraga et al. 2005. “Epigenetic differences arise during the lifetime of monozygotic twins”. ''Proceedings of the national academy of sciences of the United States of America'' 102: 10604-10609
| |
| − |
| |
| − | Gilbert, S.F., Bosch, T. M.C y Ledón-Rettig, C. 2015. “Eco-Evo-Devo: developmental symbiosis and developmental plasticity as evolutionary agents”. ''Nature Reviews Genetics'' 16: 611–622. doi:10.1038/nrg3982
| |
| − |
| |
| − | Godfrey-Smith, P. 2000. “On the theoretical role of ‛Genetic Coding’”. ''Philosophy of Science'' 67:26-44.
| |
| − |
| |
| − | Godfrey-Smith, P. 2007. “Information in Biology”. En ''The Cambridge Companion to the Philosophy of Biology, ''editado por D.'' ''Hull y M. Ruse, 103-107. Cambridge: Cambridge University Press.
| |
| − |
| |
| − | Goldberg, A. D., Allis, C. D., y Bernstein, E., 2007. “Epigenetics: a lands cape takes shape”. ''Cell'' 128( 4): 635–638.
| |
| − |
| |
| − | Griffin, H. 1999. “La clonación de Dolly”. En ''Los retos de la genética en el siglo XXI: genética y bioética, ''editado por'' ''M. Casado y R. González-Duarte, 213-213. Barcelona: Edicions Universitat de Barcelona.
| |
| − |
| |
| − | Güell, F. 2013. ''El estatuto biológico y ontológico del embrión humano: el paradigma epigenético del siglo XXI desde la teoría de la esencia de Xavier Zubiri''. Berna: Peter Lang.
| |
| − |
| |
| − | Gutarra-Vilchez, R., Santamariña-Rubio, E., Salvador, J. y Borrel, A. 2014. “Birth defects in medically assisted reproduction pregnancies in the city of Barcelona”. ''Prenatal Diagnosis'' 34:327-34.
| |
| − |
| |
| − | Hall, B. 2003. “Evo-Devo: evolutionary developmental mechanisms”. ''International Journal of Developmental Biology'' 47: 491-495.
| |
| − |
| |
| − | Hamilton W.J., Boyd, G.J., Mossman, H.W. 1945. ''Human Embryology'', 1º ed. Cambridge: W. Heffer.
| |
| − |
| |
| − | Hamilton W.J., Boyd, G.J., Mossman, H.W. 1964. ''Human Embryology'', 3º ed. Cambridge: W. Heffer & Sons.
| |
| − |
| |
| − | Hamilton W.J., Boyd, G.J., Mossman, H.W. 1972. ''Human Embryology'', 4º ed. Cambridge: W. Heffer & Sons.
| |
| − |
| |
| − | IHEC. 2016 “Research”. Consultado el 28 de julio del 2016. <nowiki>http://ihec-epigenomes.org/research/</nowiki>
| |
| − |
| |
| − | Jablonka, E. y Lamb, M.K. 2014. ''Evolution in Four Dimensions: Genetic, Epigenetic, Behavioral, and Symbolic Variation in the History of Life''. (Revised Edition). Cambridge: MIT Press.
| |
| − |
| |
| − | Jaenisch, R. y Bird, A. 2003. “Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals”. ''Nature Genetics'' 33 Suppl.: 245-254.
| |
| − |
| |
| − | Johnson, C., Warmoe, M.O., Shen, X. y Locasale, J.W. 2015. “Epigenetics and cancer metabolism”. ''Cancer Letters'' 356(2 Pt A):309-14. doi: 10.1016<nowiki>/j.ca</nowiki>nlet.2013.09.043.
| |
| − |
| |
| − | Jonas, S. y Izaurralde, E. 2015. “Towards a molecular understanding of microRNA-mediated gene silencing”. ''Nature Reviews Genetics'' 16:421–433. doi:10.1038/nrg3965
| |
| − |
| |
| − | Jones P.A. y Takai D. 2001. “The role of DNA methylation in mammalian epigenetics”. ''Science'' 293:1068–1070.
| |
| − |
| |
| − | Jouve, N. 2008. ''Explorando los genes''. Madrid: Ediciones Encuentro, S.A.
| |
| − |
| |
| − | Kim, K. et al. 2010. “Epigenetic memory in induced pluripotent stem cells”. ''Nature'' 467: 285–290.
| |
| − |
| |
| − | Kucharski, R., Maleszka, J., Foret, S. y Maleszka, R. 2008. “Nutritional control of reproductive status in honeybees via DNA methylation”. ''Science'' 319:1827-1830.
| |
| − |
| |
| − | Latschman, D.S. 2010. ''Gene control'', New York-Abingdon: Garland Science.
| |
| − |
| |
| − | Madhani, H.D., Francis, N.J., Kingston, R.E., Kornberg, R.D., Moazed, D., Narlikar, G.J., Panning B. y Struhl, K. l. 2010. “Epigenomics: A Roadmap, But to Where?”. ''Science'' 322: 43-44
| |
| − |
| |
| − | Manoj, G. T. 2016. “Micrornas in health and disease; a review of its multifaceted roles”. ''Journal of Biomedical and Pharmaceutical Research'' 5 (1): 46-51.
| |
| − |
| |
| − | Martos, S. N., Tang, W.Y. y Wang, Z. 2015. “Elusive inheritance: transgenerational effects and epigenetic inheritance in human environmental disease”. ''Progress in Biophysics and Molecular Biology'', 118(1-2):44–54.
| |
| − |
| |
| − | Mitchell, C., Schneper L.M. y Notterman, D. A. 2016. “DNA methylation, early life environment, and health outcomes”. ''Pediatric Research'' 79:212–219 doi:10.1038/pr.2015.193.
| |
| − |
| |
| − | Moczek, A.P., Sears, K.E., Stollewerk, A., Wittkopp, P. Diggle, P., Dworkin, I., Ledon-Rettig, C., Matus, D.Q., Roth, S., Abouheif, E., Brown, F.D., Chiu, C-H., Cohen, C.S., De Tomaso, A.W., Gilbert, S.F., Hall, B., Love, A.C., Lyons, D.C., Sanger, T.J., Smith, J., Specht, C., Vallejo-Marin, M. y Extavourv C.J. 2015. "The Significance and Scope of Evolutionary Developmental Biology: A Vision for the 21st Century”. ''Evolution & Development'' 17: 198–219.
| |
| − |
| |
| − | Morris, K.V, editor. 2012. ''Non-coding RNAs and Epigenetic Regulation of Gene Expression''. California: Caister Academic Press.
| |
| − |
| |
| − | Nature. 2016 “Proyecto Encode”. Consultado el 26 de julio del 2016. http:/<nowiki>/www.nature.com/encode/#</nowiki>/threads
| |
| − |
| |
| − | NIH. 2016. “The Cancer Genome Atlas”. Consultado el 28 de julio del 2016. <nowiki>http://cancergenome.nih.gov/</nowiki>
| |
| − |
| |
| − | Novo, F. J. 2016. “Human Molecular Genetics”. Consultado el 26 de julio de 2016. [http://www.unav.es/ocw/genetica/tema-1-4.html http://www.unav.es/ocw/genetica/tema-1-4.html]
| |
| − |
| |
| − | Novo, Francisco J. 2015. "Origen de la vida". En ''Diccionario Interdisciplinar Austral'', editado por Claudia E. Vanney, Ignacio Silva y Juan F. Franck URL=<nowiki>http://dia.austral.edu.ar/Origen_de_la_vida</nowiki>
| |
| − |
| |
| − | Nuño, L. 2016. “Evo-devo”. En ''Diccionario Interdisciplinar Austral'', editado por Claudia E. Vanney, Ignacio Silva y Juan F. Franck
| |
| − |
| |
| − | URL=<nowiki>http://dia.au</nowiki>stra<nowiki>l.edu.ar/Evo-devo_-_Biolologí</nowiki>a_evolutiva_del_desarrollo
| |
| − |
| |
| − | Padhee, M., Zhang, S., Lie, S., Wang, K.C., Botting, K.J., McMillen, I.C., MacLaughlin, S.M. y Morrison, J.L. 2015. “The periconceptional environment and cardiovascular disease: does in vitro embryo culture and transfer influence cardiovascular”. ''Nutrients ''7:1378–1425.
| |
| − |
| |
| − | Petronis, A. 2010. “Epigenetics as a unifying principle in the aetiology of complex traits and diseases”. ''Nature'' 465:721-727.
| |
| − |
| |
| − | Ptashne, M., Hobert, O. y Davidson, E. 2010. “Questions over the scientific basis of epigenome project”. ''Nature''. 464: 487 | doi:10.1038<nowiki>/464487c [Publicado en línea el 24 de marzo].</nowiki>
| |
| − |
| |
| − | Reik, W. 2007. “Stability and flexibility of epigenetic gene regulation in mammalian development”. ''Nature'' 447: 425–432.
| |
| − |
| |
| − | Robert, J.S. 2008. ''Embriology, epigenesis, evolution''. New York: Cambridge University Press.
| |
| − |
| |
| − | Scherrer ,U., Rexhaj, E., Allemann, Y., Sartori, C. y Rimoldi, S.F. 2015. “Cardiovascular dysfunction in children conceived by assisted reproductive technologies”. ''European Heart Journal'' 36:1583–1589.
| |
| − |
| |
| − | Stadtfeld, M., Nagaya, M., Utikal, J., Weir, G. y Hochedlinger, K. 2008. “Induced Pluripotent Stem Cells Generated Without Viral Integration”. ''Science'' 322 (5903): 945-949.
| |
| − |
| |
| − | Takahashi, K. y Yamanaka, S. 2006. “Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors”. ''Cell'' 126: 663-676.
| |
| − |
| |
| − | Tarín, J. J., García–Pérez, M. A. y Cano, A. 2014. “Assisted Reproductive Technology Results: Why Are Live–Birth Percentages So Low?”. ''Molecular Reproduction & Development'' 81:568–583.
| |
| − |
| |
| − | Watson, J. D., Baker T. A., Bell, S.P., Gann, A., Levine, M., Y Losick, R. 2008. ''Biología Molecular del Gen'' 5º ed. Madrid: Panamericana Editorial Médica.
| |
| − |
| |
| − | Watson, James D. y Crick, Francis HC. 1953. “Molecular structure of nucleic acids”. ''Nature ''171.4356:737-738.
| |
| − |
| |
| − | Wu, J., editor. 2013''. Post-Transcriptional Gene Regulation: RNA Processing in Eukaryotes''. Weinheim: Wiley-VCH Verlag GmbH & Co. KGaA.
| |
| − |
| |
| | | | |
| | ==Apéndice== | | ==Apéndice== |
| Línea 332: |
Línea 61: |
| | | | |
| | [[File:Figura 7 Epigeneticabis.jpg]] | | [[File:Figura 7 Epigeneticabis.jpg]] |
| − |
| |
| − |
| |
| − | {{Citar|url = http://dia.austral.edu.ar/Epigenética|cabecera = Güell, Francisco. 2016. "Epigenética"}}
| |
| − |
| |
| − |
| |
| − | ==Derechos de autor==
| |
| − |
| |
| − | DERECHOS RESERVADOS Diccionario Interdisciplinar Austral © Instituto de Filosofía - Universidad Austral - Claudia E. Vanney - 2016.
| |
| − |
| |
| − | ISSN: 2524-941X
| |
| − |
| |
| − |
| |
| − | ==Agradecimientos==
| |
| − |
| |
| − | Quiero agradecer al Grupo Mente-cerebro del Instituto Cultura y Sociedad de la Universidad de Navarra y y al profesor Francisco Javier Novo, también de la Universidad de Navarra, el material facilitado.
| |
| − |
| |
| − |
| |
| − | [[Categoría:Filosofía de la biología]]
| |